RNA 結合タンパク質は、DNA 修復、転写後修飾、および癌の進行など、さまざまな細胞プロセスに関与しています。自然界にはさまざまな RNA 結合タンパク質が存在しますが、新しいRNA結合ペプチドを一から設計・探索することは困難です。ELSIの研究者らが考案したスクリーニング手法は、自然界に存在しないRNA結合ペプチド配列の探索を可能にしました。
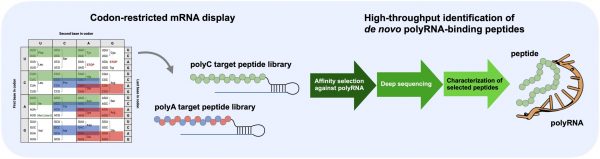
限られたアミノ酸種から成る自然界に存在しない新たなRNA 結合タンパク質の開発は、バイオテクノロジーのツールキットを拡張するだけでなく、これらの核酸とタンパク質間の相互作用を支配する基本的なメカニズム解明にもつながり、将来、人工的にRNA 結合タンパク質を設計する際の指針となります。東京工業大学 地球研究所(ELSI)の寺坂尚紘特任准教授、藤島皓介准教授、生命理工学院 生命理工学系の西川将太大学院生(博士後期課程3年)を含む研究チームは、コドンの種類を限定したDNAライブラリとmRNAディスプレイ法を用いて、一本鎖RNAに結合するペプチドを網羅的に調査しました。
mRNAディスプレイ法は、ペプチドとその遺伝情報を持つ分子(mRNA)を物理的に対応づけることで、機能性ペプチドのハイスループットな探索を可能にします。本研究では2種類の構造を取らないRNA(Poly(A) RNAとPoly(C) RNA)を標的とするペプチドライブラリを準備しました。この際、mRNA部分に含まれる塩基の種類(コドンの種類)を限定することで、標的RNAとmRNA部分の塩基対形成を防ぐ工夫を施しました。これは同時にアミノ酸の種類が限られた人工的なペプチド配列空間を探索することになります。研究チームは、約1012種類のランダムな33アミノ酸長のペプチド配列から始めて合計7ラウンドのセレクションを行いました(図1)。
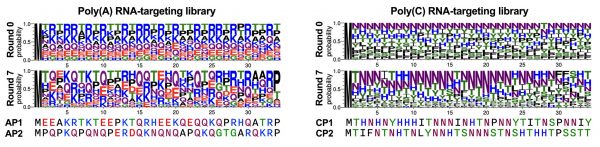
セレクション後、各ラウンドで濃縮した上位の配列を次世代シーケンサーにより明らかにすることで、一本鎖RNAに結合する4種の新規ペプチド配列を同定しました(図2)。これらのペプチド配列は特定のアミノ酸(アスパラギンまたはグルタミン)を含むモチーフが濃縮しており、それがRNA結合性に重要であることがわかりました。さらに同定したpoly(C) RNA結合ペプチドCP1は、発癌に関与する天然のRNA分子に対して強い親和性を示しました(図3)。
本手法は配列が単純な一本鎖RNAに結合可能なペプチドを同定するための強力なツールとなります。例えば生命の起源研究に関連したシンプルな核酸とタンパク質間の相互作用の理解や、将来のペプチド医薬品開発のためのアプタマーの探索など、生物学に関連した幅広い応用が可能です。

| 掲載誌 | Biomacromolecules |
| 論文タイトル | De novo single-stranded RNA-binding peptides discovered by codon-restricted mRNA display |
| 著者 | Shota Nishikawa1,2 Hidenori Watanabe1, Naohiro Terasaka1, Takayuki Katoh3, and Kosuke Fujishima1,4 |
| 所属 | 1. Earth-Life Science Institute, Tokyo Institute of Technology, Ookayama, Meguro-ku, Tokyo 152-8550, Japan 2. School of Life Science and Technology, Tokyo Institute of Technology, Ookayama, Meguro-ku, Tokyo 152-8550, Japan 3. Department of Chemistry, Graduate School of Science, The University of Tokyo, Bunkyo-ku, Tokyo 113-0033, Japan 4. Graduate School of Media and Governance, Keio University, Fujisawa 252-0882, Japan |
| DOI | 10.1021/acs.biomac.3c01024 |
| 出版日 | 2023年12月5日 |
